
EQUIPE SMol
Sondes MOLECULAIRES
Contexte scientifique et enjeux
L’équipe SMol s’intéresse à la conception, à la synthèse et à la validation de sondes moléculaires intelligentes, et envisage leur déploiement dans un large éventail d’applications incluant notamment l’imagerie de fluorescence, la photothérapie, le diagnostic précoce de maladies génétiques et l’analyse environnementale.
Un dénominateur commun est l’utilisation des techniques de fluorescence comme modalité de détection de ces objets moléculaires dans des milieux d’analyse complexes (e.g., échantillons biologiques, modèles in vivo, matrices environnementales, …). Pour atteindre ces objectifs, les membres de SMol s’appuient sur des compétences reconnues en synthèse de colorants organiques fluorescents, en photophysique moléculaire, en chimie bio(in)organique et en biologie moléculaire et cellulaire. Par ailleurs, deux membres de SMol sont également impliqués dans la plateforme d’analyses PACSMUB (mission de direction adjointe et réalisation de prestations sur des techniques spectroscopiques).
Membres de l’équipe

Responsable : Pr. Christine GOZE (PR)
- Dr. Christine STERN (MCF)
- Dr. Richard DECREAU (MCF)
- Dr. David MONCHAUD (DR CNRS)
- Marc PIROTTA (AJT UBE)
- Romain BOIDOT (IR CGFL))
- Non-permanents : liste
Axes de recherche
Axe 1 : Développement de sondes fluorescentes pour des applications en imagerie et photothérapie

En savoir plus sur nos thématiques de recherche :
Sondes fluorescentes pour la Chirurgie Guidée par Fluorescence (FGS)
La chirurgie guidée par fluorescence (FGS) est une technique innovante, notamment dans le domaine du cancer, qui permet au chirurgien de mieux visualiser certaines structures invisibles à l’œil nu, notamment des cellules tumorales, avec une précision accrue, tout en préservant au maximum l’intégrité des tissus sains. Dans ce contexte, nous travaillons sur le développement de fluorophores organiques optimisés pour la FGS, en particulier des dérivés d’aza-boro-dipyrrométhènes, qui sont des structures brillantes, chimiquement et photochimique stables, et dont les propriétés photophysiques sont adaptés pour des applications en imagerie (absorption et émission dans le proche infrarouge). Afin de les optimiser pour les applications ciblées, différentes approches moléculaires sont développées :
– Optimisation de leur solubilité dans l’eau par fonctionnalisation post-synthétique de l’atome de bore
– Décalage de leurs propriétés de fluorescence dans le NIR-II (1000-1700 nm) ce qui permet de gagner en profondeur et en sensibilité.
– Combinaison des fluorophores avec des ligands polyazamacrocycliques (agissant comme des agents chélatants de radiométaux) au sein d’une même structure, permettant de réaliser de l’imagerie bimodale et de bénéficier des avantages de chacune des techniques
– Optimisation de leurs propriétés photophysiques notamment de la durée de vie de l’état excité afin de développer des fluorophores adaptés pour l’imagerie par temps de vie, beaucoup pour sensible que l’imagerie par fluorescence.
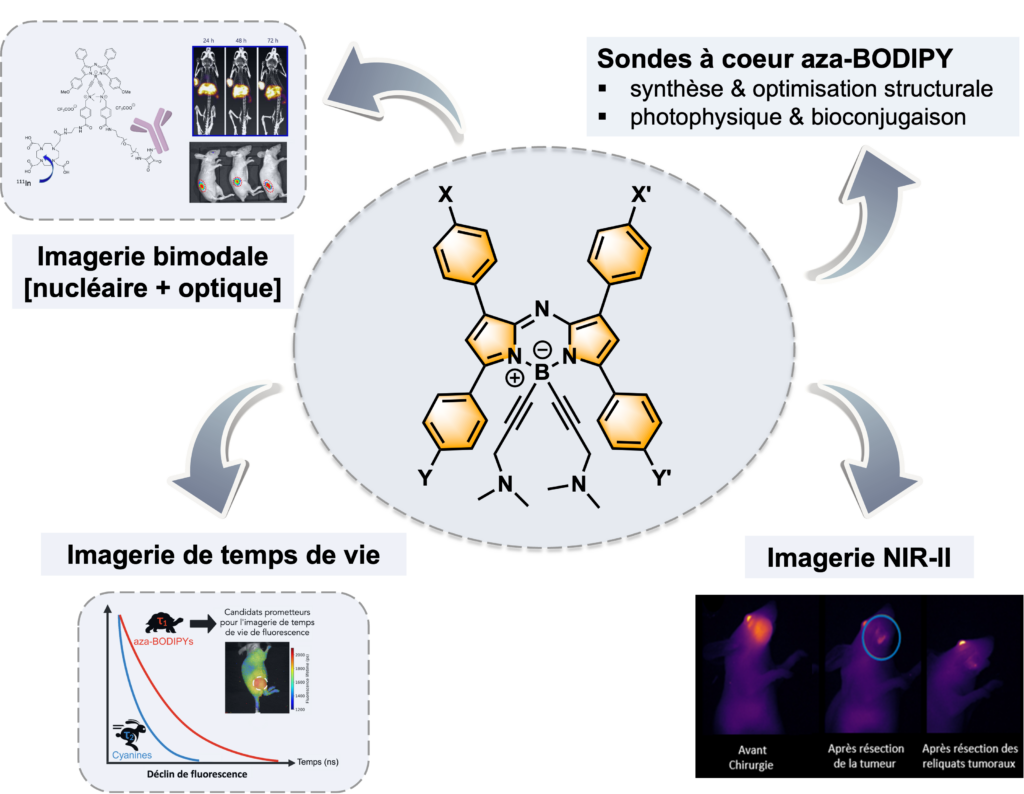
Exemples de publications de l’axe 1
- Development of an Immuno-SPECT/Fluorescent Bimodal Tracer Targeting Human or Murine PD-L1 on Preclinical Models. M. Privat, A. Massot, F. Hermetet, H. Al Sabea, C. Racoeur, N. Mabrouk, M. Cordonnier, M. Moreau, B. Collin, A. Bettaieb, F. Denat, E. Bodio, P.-S. Bellaye, C. Goze, C. Paul. Journal of Medicinal Chemistry (2024), 67, 2188-2201
- NIR-II Aza-BODIPY Dyes Bioconjugated to Monoclonal Antibody Trastuzumab for Selective Imaging of HER2-Positive Ovarian Cancer. A. Godard, G. Kalot, M. Privat, M. Bendellaa, B. Busser, K. David Wegner, F. Denat, X. Le Guével, J.-L. Coll, C. Paul, E. Bodio, C. Goze, L. Sancey. Journal of Medicinal Chemistry (2023), 66, 5185-5195
Axe 2 : Sondes fluorescentes intelligentes pour des applications en biodétection et analyse environnementales
En savoir plus sur nos thématiques de recherche :
Sondes fluorogéniques conventionnelles
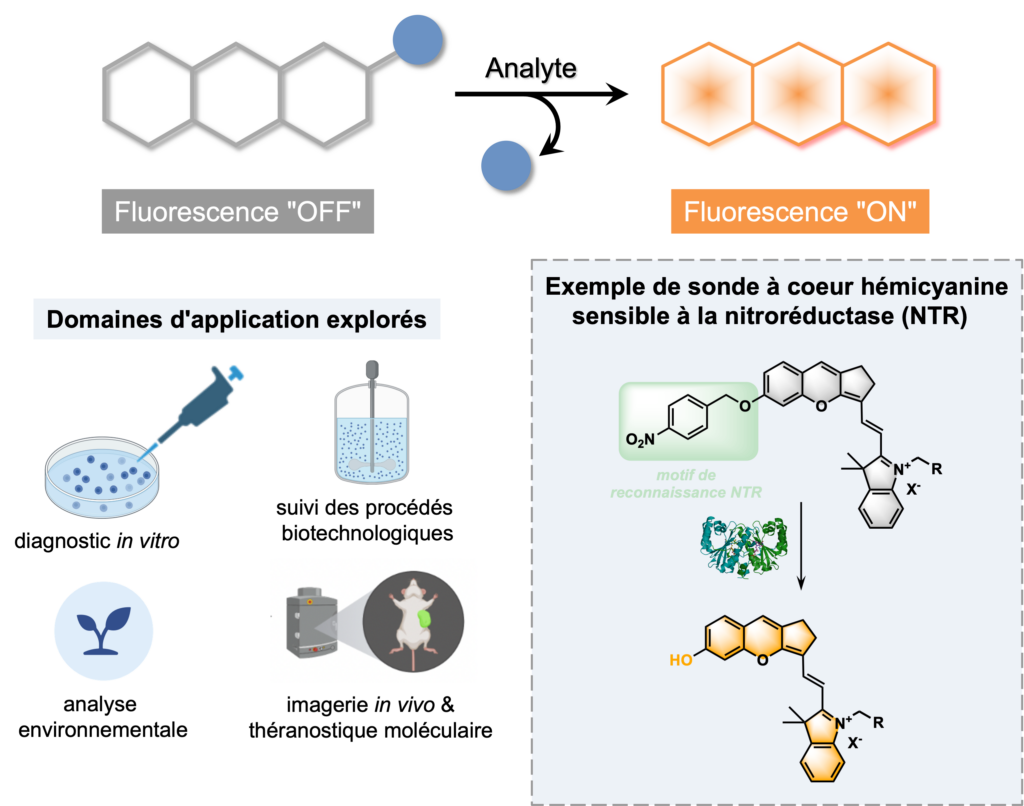
Lorsque on évoque les sondes fluorogéniques dites conventionnelles, on fait généralement référence à des fluorophores possédant un groupe fonctionnel qui va jouer le rôle d’interrupteur de fluorescence en fonction de sa substitution ou non (typiquement un groupement amino ou hydroxyle dans le cas des fluorophores à aniline ou phénol). Ainsi, lorsque la sonde réagit avec l’analyte pour lequel elle a été conçue, une réaction de déprotection du groupe fonctionnel se produit et une exaltation de fluorescence est observée (réponse « OFF-ON »). Notre contribution dans ce domaine concerne l’évaluation de nouvelles architectures photo-actives afin de concevoir des substrats fluorogéniques d’enzymes microbiennes (substrats de réductases et de peptidases). Le but étant d’accéder à des outils plus performants pour la détection bactérienne et donc potentiellement utilisables en contrôle microbiologique. Ainsi, des structures originales appartenant aux familles des dicétopyrrolopyrroles (DPPs) et des hémicyanines ont été récemment développées et évaluées notamment dans la cadre d’une collaboration de longue date avec le département R&D Microbiologie de la société bioMérieux.
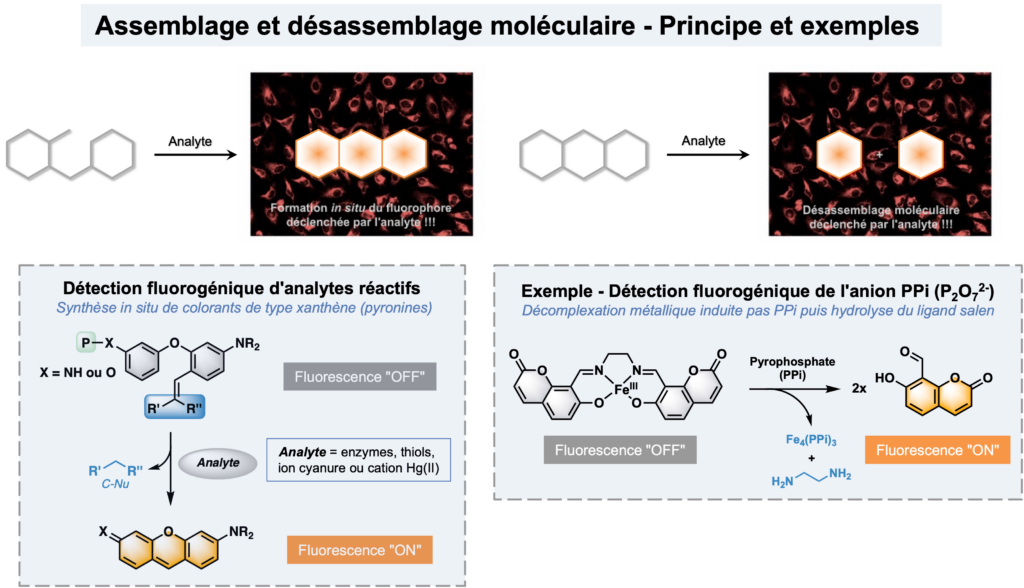
Approches moléculaires émergentes en biodétection fluorogénique : assemblage et désassemblage covalent
Afin d’accéder à des sondes moléculaires toujours plus performantes et de relever des défis actuels et à venir en (bio)analyse, nous explorons depuis une dizaine d’années une approche de rupture connue sous le nom d’assemblage covalent, particulièrement bien adaptée pour obtenir une sensibilité de détection optimale. Elle est basée sur la conception et l’utilisation de précurseurs moléculaires « cagés » qui peuvent être convertis in situ (i.e., dans le milieu d’analyse) en hétérocycles fluorescents, via des réactions domino, efficaces dans les milieux aqueux et déclenchées par l’analyte cible. Nous avons consacré plusieurs années à la mise au point, l’optimisation et la validation d’une cascade moléculaire fluorogénique originale, permettant la formation in situ de colorants pyronine brillants et émissifs au-delà de 550 nm, sous l’action d’un stimulus enzymatique. L’efficacité de ce processus domino, enzymo-déclenché a été récemment démontrée dans des cellules vivantes présentant une activité beta-galactosidase lysosomale. Cette preuve de synthèse in cellulo d’un fluorophore organique à partir d’une sonde enzymo-activable, constitue la clé de voute de nouvelles approches en (photo)théranostique moléculaire actuellement explorées au sein de notre équipe. Ce concept est également valorisé dans des applications environnementales et NRBC avec la détection de polluants (e.g., anion cyanure et cation mercurique Hg(II)) et des agents neurotoxiques organophosphorés.
Ce principe de sondes n’est toutefois applicable qu’à des analytes présentant une réactivité notable en chimie covalente. Afin de pouvoir détecter des anions d’intérêt, peu ou pas réactifs, nous avons exploré une approche alternative fondée sur des processus de décomplexation métallique et de désassemblage moléculaire récemment proposée par le groupe du Prof. Felix Zelder (Université de Zurich). Son application à la détection fluorogénique des phosphates biologiques est actuellement envisagée.
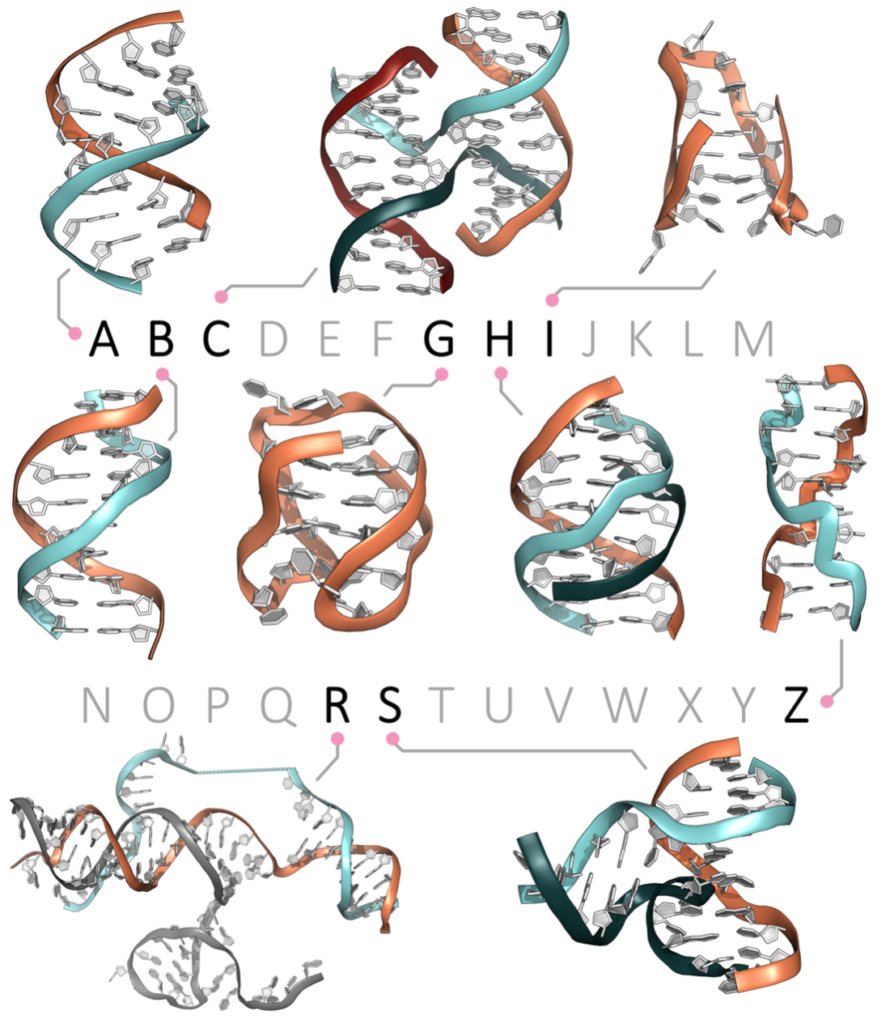
Des ligands multifonctionnels pour les structures alternatives des acides nucléiques
Notre société a besoin de nouvelles pistes thérapeutiques pour faire face au vieillissement global de la population et à la croissance inexorable des maladies qui y sont afférentes, notamment les cancers et les maladies neurodégénératives. Les récents progrès thérapeutiques ont été rendus possibles par le développement du séquençage, qui a permis un génotypage toujours plus précis des maladies génétiques et donc, l’identification de marqueurs à fort potentiel stratégique. Ces avancées ouvrent la voie à un ciblage précis des dérégulations génétiques sous-jacentes à ces maladies, et donc, à l’avènement d’une médecine de précision. Toutefois, le séquençage ne permet que l’identification des cibles génétiques et, à ce jour, le ciblage sélectif de séquences d’ADN avec des petites molécules n’est toujours pas une réalité. Une alternative au ciblage de séquences d’ADN connaît actuellement une grande popularité : le ciblage des structures de l’ADN, et notamment les structures alternatives de l’ADN. En effet, au-delà de la double hélice (ou duplexe), l’ADN peut revêtir localement différentes structures comprenant non pas deux (comme le duplexe) mais trois ou quatre brins (comme les triplexes (H-DNA), les jonctions d’ADN (C- et S-DNA), les quadruplexes (G- et I-DNA), etc.). Ces édifices possèdent des structures tridimensionnelles bien plus riches que les duplexes, ce qui permet d’envisager un ciblage plus précis. Au cours des dernières années, notre contribution dans ce domaine a été multiple : nous avons notamment montré que les quadruplexes et les jonctions d’ADN à trois voies, jouent des rôles importants dans les mécanismes pathologiques liés aux cancers et maladies neurodégénératives. Nous avons aussi développé des outils moléculaires intelligents, qui sont utilisés comme ligands, sondes imageantes et hameçons moléculaires pour gagner des informations toujours plus précises sur le rôle de ces structures dans les dérégulations génétiques à l’origine de ces pathologies.
Exemples de publications de l’axe 2
- Nanopore adaptive sampling accurately detects nucleotide variants and improves the characterization of large-scale rearrangement for the diagnosis of cancer predisposition. S. Chevrier, C. Richard, M. Mille, D. Bertrand, R. Boidot. Clinical Translation Medicine (2025), 15, e70138
- Clinical evaluation of a low-coverage whole-genome test for detecting homologous recombination deficiency in ovarian cancer. R. Boidot, M. G. B. Blum, M.-P. Wissler, C. Gottin, J. Ruzicka, S. Chevrier, T. M. Delhomme, J. Audoux, A. Jeanniard , P.-A. Just, P. Harter, S. Pignat, A. González-Martin, C. Marth, J. Mäenpää, N. Colombo, I. Vergote, K. Fujiwara, N. Duforet-Frebourg, D. Bertrand, N. Philippe, I. Ray-Coquard, E. Pujade-Lauraine, PAOLA-1/ ENGOT-ov25 Study Group.European Journal of Cancer (2024), 202, 113978
- Durable response of lung carcinoma patients to EGFR tyrosine kinase inhibitors is determined by germline polymorphisms in some immune-related genes. L. Dalens, J. Niogret, C. Richard, S. Chevrier, P. Foucher, B. Coudert, A. Lagrange, L. Favier, V. Westeel, S. Kim, O. Adotevi, C. Chapusot, L. Martin, L. Arnould, C.-G. Kaderbhai, R. Boidot. Molecular Cancer (2023), 22, 120
Axe 3 : Développement technologique en biologie moléculaire

En savoir plus sur nos thématiques de recherche :
Biologie moléculaire et séquençage
L’unité de Biologie Moléculaire du CGFL est un laboratoire de diagnostic moléculaire de routine. Cependant, il possède une activité de recherche axée sur le développement technologique basé sur les nouvelles technologies de séquençage ainsi que sur la recherche de biomarqueurs.
Le laboratoire met notamment l’accent sur le séquençage de 3ème génération par nanopores qui permet de séquencer des acides nucléiques natifs, c’est-à-dire tel qu’ils sont présents dans les cellules. La première application développée et publiée en janvier 2025 a démontré l’utilisation de ce type de séquençage pour la détection des altérations responsables de la prédisposition génétiques aux cancers dont celui du sein, de l’ovaire, de l’appareil digestif, du pancréas et de la prostate.
Actuellement, le laboratoire a deux projets en cours :
– Développer un kit de détection rapide des altérations des gènes de prédisposition au cancer du sein pour rendre un résultat en 2 jours au lieu de 3 à 6 mois.
– Etudier l’ADN tumoral circulant pour le monitoring, le suivi de la maladie résiduelle et le dépistage des cancers solides à partir d’une simple prise de sang.
Le laboratoire est également ouvert à tout développement permettant à des partenaires d’enrichir leurs recherches avec des techniques de séquençage
Exemples de publications de l’axe 3
- Nanopore adaptive sampling accurately detects nucleotide variants and improves the characterization of large-scale rearrangement for the diagnosis of cancer predisposition. S. Chevrier, C. Richard, M. Mille, D. Bertrand, R. Boidot. Clinical Translation Medicine (2025), 15, e70138
- Clinical evaluation of a low-coverage whole-genome test for detecting homologous recombination deficiency in ovarian cancer. R. Boidot, M. G. B. Blum, M.-P. Wissler, C. Gottin, J. Ruzicka, S. Chevrier, T. M. Delhomme, J. Audoux, A. Jeanniard , P.-A. Just, P. Harter, S. Pignat, A. González-Martin, C. Marth, J. Mäenpää, N. Colombo, I. Vergote, K. Fujiwara, N. Duforet-Frebourg, D. Bertrand, N. Philippe, I. Ray-Coquard, E. Pujade-Lauraine, PAOLA-1/ ENGOT-ov25 Study Group.European Journal of Cancer (2024), 202, 113978
- Durable response of lung carcinoma patients to EGFR tyrosine kinase inhibitors is determined by germline polymorphisms in some immune-related genes. L. Dalens, J. Niogret, C. Richard, S. Chevrier, P. Foucher, B. Coudert, A. Lagrange, L. Favier, V. Westeel, S. Kim, O. Adotevi, C. Chapusot, L. Martin, L. Arnould, C.-G. Kaderbhai, R. Boidot. Molecular Cancer (2023), 22, 120
.
Projets en cours
Nous bénéficions du soutien de diverses agences de financement (ANR, Ligue contre le Cancer, MITI CNRS, programme Horizon Europe, …) et de la région BFC via les fonds FEDER et la Graduate School INTHERAPI. Une part de nos activités est également consacrée à des actions de valorisation conduites avec le soutien de la SATT SAYENS, de CNRS Innovation ou en partenariat avec des jeunes pousses (CHEMFORASE, FLUONIR, SON) ou des sociétés de renommée internationale (Merck KGaA et Strem Inc.).